Segment anything in medical images
Abstract
医学图像分割是临床实践的重要组成部分,有助于准确诊断,治疗计划和疾病监测。然而,现有的方法往往是针对特定的模式或疾病类型量身定制的,缺乏在医学图像分割任务的各种范围内的通用性。在这里,我们提出MedSAM,一个基础模型,旨在通过实现通用医学图像分割来弥合这一差距。该模型是在一个大型医学图像数据集上开发的,该数据集包含1,570,263个图像mask对,涵盖10种成像方式和30多种癌症类型。我们对86个内部验证任务和60个外部验证任务进行了综合评估,证明了比模态专家模型更好的准确性和鲁棒性。通过在广泛的任务范围内提供准确有效的细分,MedSAM在加速诊断工具的发展和治疗计划的个性化方面具有巨大的潜力。
分割是医学成像分析中的一项基本任务,涉及识别和描绘各种医学图像(如器官、病变和组织)中的感兴趣区域(ROI) 1。准确的分割对于许多临床应用至关重要,包括疾病诊断、治疗计划和疾病进展监测2,3。人工分割长期以来一直是描绘解剖结构和病理区域的黄金标准,但这一过程耗时,劳动密集,并且通常需要高度的专业知识。半自动或全自动分割方法可以显著减少所需的时间和人工,提高一致性,并能够分析大规模数据集。
基于深度学习的模型在医学图像分割中显示出巨大的前景,因为它们能够学习复杂的图像特征,并在各种任务中提供准确的分割结果,从分割特定的解剖结构到识别病理区域。然而,当前许多医学图像分割模型的一个显著限制是它们的任务特异性。这些模型通常是为特定的分割任务设计和训练的,当应用于新任务或不同类型的成像数据时,它们的性能可能会显著下降。这种通用性的缺乏对这些模型在临床实践中的广泛应用构成了实质性的障碍。作为对比,在自然图像分割的最新进展已经见证了分割基础模型的涌现,如分割任何模型(SAM)和分割任何地方与多模态提示同时出现,在各种分割任务中展示了卓越的多功能性和性能。
医学图像分割中对通用模型的需求越来越大:可以训练一次然后应用于广泛的分割任务的模型。这样的模型不仅在模型能力方面表现出更高的通用性,而且还可能导致跨不同任务的更一致的结果。然而,由于自然图像与医学图像的显著差异,分割基础模型(如SAM)在医学图像分割中的适用性仍然有限。本质上,SAM是一种提示分割方法,它需要点或边界框来指定分割目标。这与传统的交互式分割方法类似,但SAM具有更好的泛化能力,而现有的基于深度学习的交互式分割方法主要集中在有限的任务和图像模态上。
许多研究已经将开箱即用的SAM模型应用于典型的医学图像分割任务(12 - 17)和其他具有挑战性的场景(18 - 21)。例如,同时进行的研究对不同医学图像阵列的SAM实施了综合评估,强调SAM主要在具有明显边界特征的目标上取得了令人满意的分割结果。然而,该模型在分割弱边界或低对比度的典型医学目标时存在很大的局限性。根据这些观察结果,我们进一步引入了MedSAM,这是一种改进的基础模型,可以显著提高SAM对医学图像的分割性能。MedSAM通过在一个前所未有的数据集上对SAM进行微调来实现这一目标,该数据集拥有超过100万个医学图像掩码对。
我们通过86项内部验证任务和60项外部验证任务的综合实验,涵盖了各种解剖结构、病理条件和医学成像方式,对MedSAM进行了全面的评估。实验结果表明,MedSAM始终优于最先进的(SOTA)分割基础模型,同时实现了与来自相同模态的图像训练的专家模型1,24相当的性能,甚至超过了专家模型1,24。这些结果突出了MedSAM作为多功能医学图像分割新范例的潜力。
Results
MedSAM: a foundation model for promptable medical image segmentation
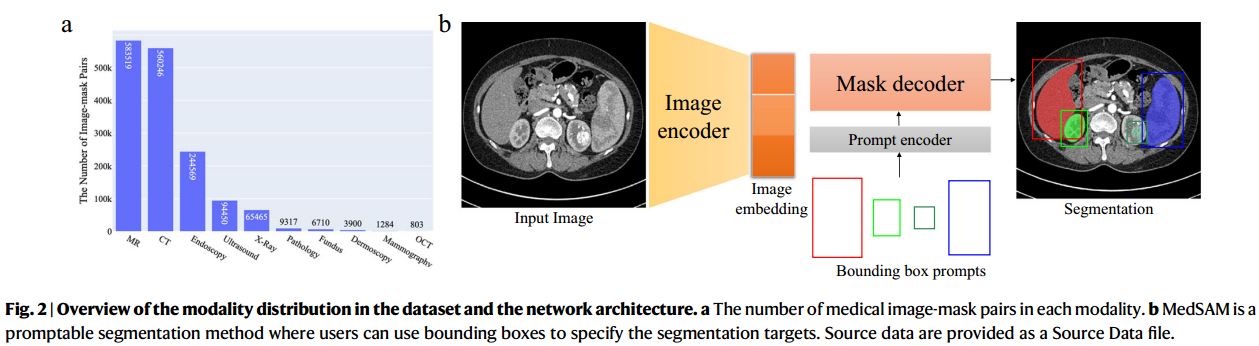
MedSAM旨在实现通用医学图像分割的基础模型的作用。构建这种模型的一个关键方面是适应成像条件、解剖结构和病理条件的广泛变化的能力。为了应对这一挑战,我们制作了一个多样的和大规模医学图像分割数据集,包含1,570,263个医学图像掩模对,涵盖10种成像方式,超过30种癌症类型和多种成像方案(图1和补充表1 - 4)。这个大规模的数据集允许MedSAM学习丰富的医学图像表示,捕获不同模式的广泛解剖学和病变。图2a概述了数据集中不同医学成像模式的图像分布,并按其总数进行了排名。很明显,计算机断层扫描(CT)、磁共振成像(MRI)和内窥镜检查是主要的检查方式,反映了它们在临床实践中的普遍性。CT和MRI图像提供三维身体结构的详细横断面视图,使其成为非侵入性诊断成像不可或缺的工具。虽然内窥镜检查更具侵入性,但它可以对器官内部进行直接的视觉检查,对胃肠道和泌尿系统疾病的诊断证明是非常宝贵的。尽管这些方法很流行,但其他方法如超声、病理、眼底、皮肤镜、乳房x光检查和光学相干断层扫描(OCT)在临床实践中也发挥着重要作用。这些模式及其相应的分割目标的多样性强调了能够处理与每种模式相关的独特特征的通用和有效的分割模型的必要性。
另一个重要的考虑因素是选择适当的分段提示符和网络体系结构。虽然全自动分割基础模型的概念很诱人,但它充满了使其不切实际的挑战。主要的挑战之一是分割任务固有的可变性。例如,给定肝癌CT图像,分割任务可以根据具体的临床情况而变化。一个临床医生可能会对分割肝脏肿瘤感兴趣,而另一个可能需要分割整个肝脏和周围器官。此外,成像方式的可变性提出了另一个挑战。CT和MR等方式产生3D图像,而x射线和超声波等方式产生2D图像。任务定义和成像模式的这些变化使得能够准确预测和解决不同用户不同需求的全自动模型的设计复杂化。
考虑到这些挑战,我们认为一个更实用的方法是开发一个快速的二维分割模型。该模型可以根据用户提供的提示轻松地适应特定任务,从而提供增强的灵活性和适应性。它还能够通过将3D图像处理为一系列2D切片来处理2D和3D图像。典型的用户提示包括点和边界框,我们在补充图1中展示了一些使用不同提示的分割示例。可以发现,边界框为感兴趣的区域提供了更明确的空间背景,使算法能够更精确地识别目标区域。这与基于点的提示形成鲜明对比,后者可能会引入歧义,特别是当近似结构彼此相似时。此外,绘制边界框是有效的,特别是在涉及多目标分割的场景中。我们遵循SAM中的网络架构,包括一个图像编码器、一个提示编码器和一个掩码解码器(图2b)。图像编码器25将输入图像映射到高维图像嵌入空间。提示编码器通过位置编码将用户绘制的边界框转换为特征表示26。最后,掩码解码器使用交叉关注融合图像嵌入和提示特征27(方法)。

Quantitative and qualitative analysis
我们通过内部验证和外部验证来评估MedSAM。具体来说,我们将其与SOTA分割基础模型SAM7以及模态专家U-Net1和DeepLabV3+24模型进行了比较。每个专门的模型都在相应模态的图像上进行训练,从而为每种方法生成10个专门的专家模型。在推理过程中,使用这些专家模型从相应的模态中分割图像,而使用SAM和MedSAM对所有模态的图像进行分割(方法)。内部验证包含86个分割任务(补充表5-8和图2),图3a显示了四种方法的这些任务的中位数骰子相似系数(DSC)得分。总体而言,SAM在大多数分割任务中获得了最低的性能,尽管它在一些RGB图像分割任务中表现良好,例如内镜图像中的息肉(DSC: 91.3%,四分位间距(IQR): 81.2-95.1%)分割。这可能是由于SAM在各种RGB图像上进行了训练,并且这些图像中的许多目标由于其不同的外观而相对容易分割。其他三种模型的表现明显优于SAM,并且MedSAM在86个区间验证任务的DSC分数分布比两组专家模型更窄,反映了MedSAM在不同任务中的稳健性。我们进一步将四种模型的同一任务对应的DSC分数与图3b的讲台图联系起来,与箱形图相辅相成。在上半部分,每个彩色点表示用各自的方法在一个任务上获得的DSC中值。对应于相同测试用例的点由一条线连接起来。在下面的部分,每种方法获得排名的频率用柱状图表示。可以发现,MedSAM在大多数任务中排名第一,超过了U-Net和DeepLabV3+专家模型的表现,后者分别排名第二和第三,排名频率很高,相比之下,SAM在几乎所有任务中排名最后。图3c(及补充图9)显示了随机选取的MedSAM获得DSC中位数评分的分割示例,包括CT图像中的肝肿瘤、MR图像中的脑肿瘤、超声图像中的乳腺肿瘤和内镜图像中的息肉。SAM与边界较弱的目标作斗争,容易出现分割不足或分割过度错误。相比之下,MedSAM可以在各种成像条件下准确地分割大范围的目标,甚至比专业的U-Net和DeepLabV3+模型更好。
外部验证包括60个分割任务,这些任务要么来自新的数据集,要么涉及未见过的分割目标(补充表9-11和图10-12)。图4a、b分别显示了60个任务的DSC分数中位数分布及其对应关系。尽管SAM在大多数CT和MR分割任务上继续表现较低,但专家模型的表现不再优于SAM(例如,在MR t1加权图像中,SAM、U-Net和DeepLabV3+的右肾分割率分别为90.1%、85.3%和86.4%)。这表明这种专家模型对未知目标的泛化能力有限。相比之下,MedSAM始终提供卓越的性能。例如,MedSAM在鼻咽癌分割任务上的DSC中位数得分为87.8% (IQR: 85.0-91.4%),比SAM、专家U-Net和DeepLabV3+分别提高52.3%、15.5%和22.7分。值得注意的是,MedSAM在一些不可见的模式(例如腹部T1 Inphase和Outphase)中也取得了更好的性能,超过SAM和专家模型,改进高达10%。图4c给出了四个随机选择的分割示例进行定性评价,结果表明,虽然所有方法都具有处理简单分割目标的能力,但MedSAM在分割具有难以区分边界的挑战性目标方面表现更好,例如MR图像中的宫颈癌(更多示例见补充图13)。此外,我们在多发性骨髓瘤浆细胞数据集上评估了MedSAM,与之前所有的验证任务相比,它代表了一种独特的模式和任务。尽管该任务在训练过程中从来没被看见,




MedSAM仍然表现出优于SAM的性能(Supplementary Fig. 14),突出了其出色的泛化能力。
The effect of training dataset size
我们还研究了不同数据集大小对MedSAM性能的影响,因为训练数据集大小已被证明是模型性能的关键28。我们还在两种不同的数据集大小上训练MedSAM: 10,000 (10K)和100,000 (100K)图像,并将其性能与默认MedSAM模型进行比较。10K和100K的训练图像从整个训练集中均匀采样,保持数据的多样性。如图5a所示(补充表12-14),性能遵循缩放规则,增加训练图像的数量可以显著提高内部和外部验证集的性能。
MedSAM can improve the annotation efficiency
此外,我们进行了一项人工注释研究,以评估两种管道的时间成本(方法)。对于第一条流水线,两名人类专家以逐片的方式手动注释3D肾上腺肿瘤。对于第二条流水线,专家们首先每3-10片用线性标记(初始标记)画出肿瘤的长、短轴,这是肿瘤反应评价中常见的做法。然后,利用MedSAM基于这些稀疏线性注释对肿瘤进行分割。最后,由专家手动修改分割结果,直到满意为止。我们定量地比较了两种管道之间的标注时间成本(图5b)。结果表明,在MedSAM的辅助下,两位专家的标注时间分别大幅缩短了82.37%和82.95%。
Discussion
我们介绍MedSAM,这是一种基于深度学习的基础模型,用于在不同的医学成像模式下对大量解剖结构和病变进行分割。MedSAM是在一个精心组装的大型数据集上进行训练的,该数据集由超过一百万的医学图像掩模对组成。它的快速配置在自动化和定制之间取得了最佳平衡,使MedSAM成为通用医学图像分割的通用工具。
通过包括内部和外部验证在内的综合评估,MedSAM已经展示了在分割不同目标阵列和管理新数据和任务的强大泛化能力方面的实质性能力。其性能不仅明显超过现有最先进的细分基础模型,而且可以与专业模型相媲美甚至超越。通过提供解剖结构和病理区域的精确描述,MedSAM促进了作为生物标志物的各种定量测量的计算。例如,MedSAM可以在加速3D肿瘤注释过程中发挥关键作用,从而实现肿瘤体积的后续计算,这是评估疾病进展和治疗反应的关键生物标志物29。此外,MedSAM为将自然图像基础模型应用于新领域提供了一个成功的范例,这可以进一步扩展到生物图像分割30,例如光学显微镜图像中的细胞分割31和电子显微镜图像中的细胞器分割3。
虽然MedSAM拥有强大的能力,但它也存在一定的局限性。其中一个限制是训练集中的模态不平衡,CT、MRI和内窥镜图像在数据集中占主导地位。这可能会潜在地影响模型在代表性较低的模式上的表现,比如乳房x光检查。另一个限制是它在分割血管状分支结构方面的困难,因为在这种情况下,边界框提示符可能是模糊的。例如,在眼底图像中,动脉和静脉共享同一个边界框。然而,这些限制并没有减少MedSAM的效用。由于MedSAM从大规模训练集中学习了丰富且具有代表性的医学图像特征,因此可以对其进行微调,以有效地从代表性较低的模式或复杂的结构(如血管)中分割新任务。
总之,本研究强调了构建一个能够管理大量分割任务的单一基础模型的可行性,从而消除了对任务特定模型的需求。MedSAM作为医学图像分割的首个基础模型,在加速新诊断和治疗工具的发展,并最终为改善患者护理做出贡献方面具有巨大潜力3
Methods
Dataset curation and pre-processing
我们通过整理来自互联网上各种来源的公开医学图像分割数据集的图像来策划一个全面的数据集,这些数据集包括癌症成像档案(TCIA)34、Kaggle、Grand-Challenge、科学数据、CodaLab和医学图像计算和计算机辅助干预协会(MICCAI)的分割挑战。所有数据集都提供了人类专家的分割注释,这些注释在现有文献中已经被广泛使用(补充表1-4)。我们将这些注释直接合并到模型开发和验证中。
原始的3D数据集包括DICOM、nrrd或mhd格式的计算机断层扫描(CT)和磁共振(MR)图像。为了确保医学图像深度学习模型的一致性和兼容性,我们将图像转换为广泛使用的NifTI格式。此外,将灰度图像(如x射线和超声)以及RGB图像(包括内窥镜、皮肤镜、眼底和病理图像)转换为png格式。

为了提高数据集的质量和一致性,采用了几个独特的标准,包括不完整的图像和带有分支结构的分割目标、不准确的注释和微小的体积。值得注意的是,不同模态的图像强度差异很大。例如,CT图像的强度值范围为-2000至2000,而MR图像的强度值范围为0至3000。在内窥镜和超声图像中,强度值通常从0到255。为了促进稳定的训练,我们对所有图像执行强度归一化,确保它们共享相同的强度范围。
对于CT图像,我们最初使用典型窗宽和水平值对Hounsfield单元进行归一化。软组织、肺和脑的窗宽和水平值分别为(W:400, L:40)、(W:1500, L:-160)和(W:80, L:40)。随后,将强度值重新缩放到[0,255]的范围。对于MR, x射线,超声,乳房x光摄影和光学相干断层扫描(OCT)图像,我们将强度值裁剪到0.5到99.5百分位数之间,然后将其重新缩放到[0,255]的范围。对于RGB图像(如内窥镜、皮肤镜、眼底和病理图像),如果已经在预期的强度范围内[0,255],则其强度保持不变。但是,如果它们超出了这个范围,我们使用最大最小归一化将强度值重新缩放为[0,255]。最后,为了满足模型的输入要求,将所有图像调整为1024 × 1024 × 3的统一尺寸。在全片病理图像的情况下,使用滑动窗口方法提取斑块,没有重叠。位于边界上的补丁被填充为0。对于3D CT和MR图像,将每个2D切片调整为1024 × 1024,通道重复三次以保持一致性。其余2D图像直接调整为1024 × 1024 × 3。采用双三次插值法调整图像大小,采用最近邻插值法调整蒙版大小,以保持其精确边界,避免引入不必要的伪影。这些标准化程序确保了所有图像的统一性和兼容性,并促进了无缝集成到模型训练和评估管道的后续阶段。
Network Structure
本研究中使用的网络建立在transformer架构上27,该架构在自然语言处理和图像识别任务等各个领域显示出显著的有效性25。具体来说,该网络包含了一个基于视觉转换器(ViT)的图像编码器,负责提取图像特征,一个用于集成用户交互(边界框)的提示编码器,以及一个使用图像嵌入、提示嵌入和输出令牌生成分割结果和置信度分数的掩码解码器。
为了在分割性能和计算效率之间取得平衡,我们采用基本的ViT模型作为图像编码器,因为广泛的评估表明,更大的ViT模型,如ViT Large和ViT Huge,在精度上只提供了微小的改进,而大大增加了计算需求。具体来说,基本ViT模型由12个变压器层组成27,每个块包括一个多头自关注块和一个多层感知器(MLP)块,其中包含层归一化35。使用掩码自编码器建模进行预训练36,然后在SAM数据集上进行完全监督训练7。将输入图像(1024 × 1024 × 3)重构为一系列尺寸为16 × 16 × 3的平面二维小块,经过图像编码器后得到图像嵌入中的特征尺寸为64 × 64,缩小了16倍。提示编码器将边界框提示符的角点映射到256维向量嵌入26。其中,每个边界框由左上角点和右下角点的嵌入对表示。为了方便实时用户交互,一旦图像嵌入计算完成,采用一个轻量级掩码解码器架构。它由两个transformer层27和两个转置卷积层组成,前者用于融合图像嵌入和提示编码,后者用于将嵌入分辨率提高到256 × 256。随后,对嵌入进行s型激活,然后进行双线性插值以匹配输入大小。
Training protocol and experimental setting
在数据预处理过程中,我们获得了1,570,263对医学图像掩模,用于模型开发和验证。对于内部验证,我们将数据集随机分成80%、10%和10%,分别作为训练、调优和验证。具体来说,对于扫描内存在连续性的模式,如CT和MRI,以及连续帧之间存在连续性的模式,我们分别在3D扫描和视频级别执行数据分割,从而防止任何潜在的数据泄漏。对于病理图像,认识到幻灯片级内聚性的重要性,我们首先将整个幻灯片图像分离为不同的基于幻灯片的图像集。然后将每张幻灯片分成固定大小为1024 × 1024的小块。这种设置允许我们监视模型在调谐集上的性能,并在训练期间调整其参数以防止过拟合。对于外部验证,所有数据集都被保留,并且在模型训练期间不出现。这些数据集为模型的泛化能力提供了严格的测试,因为它们代表了模型以前没有遇到过的新患者、成像条件和潜在的新分割任务。通过评估MedSAM在这些看不见的数据集上的表现,我们可以对MedSAM在现实世界的临床环境中可能的表现有一个现实的理解,在现实世界中,MedSAM需要处理数据中的大量可变性和不可预测性。训练和验证是独立的。
使用预训练的SAM模型和ViT-Base模型对模型进行初始化。我们修复了提示编码器,因为它已经可以对边界框提示进行编码。在训练过程中更新图像编码器和掩码解码器中的所有可训练参数。具体来说,图像编码器和掩码解码器的可训练参数数量分别为89,670,912和4,058,340。用0 ~ 20像素的随机扰动从专家注释中模拟边界框提示。损失函数是骰子损失和交叉熵损失的未加权和,已被证明在各种分割任务中具有鲁棒性1。采用AdamW37优化器(β1 = 0.9, β2 = 0.999)对网络进行优化,初始学习率为1e-4,权值衰减为0.01。全局批大小为160,没有使用数据增强。模型在20个A100 (80G) gpu上进行150个epoch的训练,最后一个检查点作为最终模型。
此外,为了全面评估MedSAM的性能,我们对最先进的分割基础模型SAM7和专家模型(即U-Net1和DeepLabV3+24)进行了比较分析。训练图像包含10种模式:CT、MR、胸部x光(CXR)、皮肤镜、内窥镜、超声、乳房x光、OCT和病理,我们针对每种模式训练了U-Net和DeepLabV3+专家模型。专家模型共有20个,对应的训练图像数量见补充表5。我们使用nnU-Net进行所有U-Net实验,它可以根据数据集属性自动配置网络架构。为了将边界框提示合并到模型中,我们将边界框转换为二进制掩码,并将其与图像连接作为模型输入。该功能最初是由级联管道中的nnU-Net支持的,通过使用二进制掩码作为指定目标位置的附加通道,在许多分段任务中证明了提高的性能。训练设置遵循2D nnU-Net的默认配置。每个模型在一个A100 GPU上进行1000个epoch的训练,最后一个检查点作为最终模型。DeepLabV3+专家模型使用ResNet5038作为编码器。与参考文献3类似,输入图像被调整为224 × 224 × 3。将边界框转换为二进制掩码作为额外的输入通道,以提供对象位置提示。使用Pytorch(0.3.3)39对所有模态专家DeepLabV3 +模型进行训练和推理。每个模态智能模型在一个A100 GPU上进行500个epoch的训练,最后一个检查点作为最终模型。在推理阶段,使用SAM和MedSAM对单个模型的所有模态进行分割。相比之下,U-Net和DeepLabV3+专家模型被用于单独分割各自对应的模式。
对于某些应用程序,特定于任务的分段模型可能优于基于模态的分段模型。由于U-Net在大多数任务上的表现都优于DeepLabV3+,我们进一步通过训练特定任务的U-Net模型,对CT扫描中的肝癌分割、MR扫描中的腹部器官分割、超声图像中的神经癌分割、内镜图像中的息肉分割等4个代表性任务进行对比研究。实验包括内部验证和外部验证。
对于内部验证,我们坚持默认的数据分割,使用它们来训练特定于任务的U-Net模型,然后在相应的验证集上评估它们的性能。对于外部验证,训练好的U-Net模型在来自相同模态或分割目标的新数据集上进行评估。在所有这些实验中,MedSAM都直接应用于验证集,没有额外的微调。如图15所示,虽然特定于任务的UNet模型通常在内部验证集上取得了很好的结果,但它们的性能在外部验证集上显著下降。相比之下,MedSAM在内部和外部验证集上都保持一致的性能。这强调了MedSAM优越的泛化能力,使其成为各种医学图像分割任务的通用工具。
Loss function
我们使用交叉熵损失和骰子损失之间的未加权和40作为最终损失函数,因为它已被证明在不同的医学图像分割任务中具有鲁棒性41。其中,S, G分别表示分割结果和ground truth。Si, gi分别表示体素i的预测分割和ground truth。N是图像i中的体素数,二值交叉熵损失定义为:

dice loss被定义为:

最终损失L定义为:

Human annotation study
人类注释研究的目的是定量评估MedSAM如何减少注释时间成本。具体来说,我们使用了最近的肾上腺皮质癌CT数据集34,42,43,其中分割目标肾上腺肿瘤既不是训练的一部分,也不是现有验证集的一部分。我们随机抽取10例,共733个需要注释的肿瘤切片。两位人类专家参与了这项研究,他们分别有8年和6年腹部疾病临床实践经验的放射科医生,每位专家生成了两组注释,一组有MedSAM的帮助,另一组没有。
在第一组中,专家们以逐片的方式手工标注3D肾上腺肿瘤。两位专家的注释是独立进行的,没有协作讨论,并且记录了每个案例所花费的时间。在第二组中,经过一周的冷却期后生成注释。专家独立绘制肿瘤长轴和短轴作为初始标记,这是肿瘤反应评价的常见做法。这个过程从肿瘤的顶部切片到底部切片每3-10片执行一次。然后,我们基于这些稀疏线性注释应用MedSAM对肿瘤进行分割,分为三个步骤。
- 步骤1。对于每个标注的切片,基于线性标签生成一个可以完全覆盖线性标签的矩形二值掩码。
- 步骤2。对于未标记的切片,通过对周围标记的切片进行插值生成矩形二值蒙版。
- 步骤3。我们将二值蒙版转换为边界框,然后将其与图像一起馈送到MedSAM中生成分割结果。
所有这些步骤都是自动进行的,并记录每种情况下的模型运行时间。最后,由人类专家对分割结果进行人工细化,直到达到自己满意的程度。总而言之,第二组注释的时间成本包含三个部分:初始标记、MedSAM推理和细化。所有手工标注过程均基于ITKSNAP44,这是一款为医学图像可视化和标注而设计的开源软件。
Evaluation metrics
我们遵循Metrics reloadd45中的建议,使用骰子相似系数和归一化表面距离(NSD)来定量评估分割结果。DSC是一种基于区域的分割度量,旨在评估专家标注掩码与分割结果之间的区域重叠,定义为:

NSD46是一种基于边界的度量,旨在评估专家标注掩码和分割结果在给定容限下的边界一致性,该容限由:

其中
 分别表示专家标注掩码的边界区域和公差τ处的分割面。在本文中,我们设公差τ为2。
分别表示专家标注掩码的边界区域和公差τ处的分割面。在本文中,我们设公差τ为2。
Statistics analysis
为了统计分析和比较上述四种方法(MedSAM、SAM、U-Net和DeepLabV3+专家模型)的性能,我们采用了Wilcoxon符号秩检验。这种非参数检验非常适合比较成对样本,当数据不符合正态分布假设时特别有用。该分析使我们能够确定是否有任何方法在统计上表现出优于其他方法的分割性能,从而为评估方法的比较有效性提供有价值的见解。Wilcoxon sign -rank检验结果在DSC和NSD计分表上进行标注(补充表6-11)。
